(Adaptado de lab dgolombek/ssimonetta. UNQ/FIL 2013)
Link Archivo: Protocolos.PDF
- NGM (Agar de crecimiento de gusanos)
- LB (medio para cultivo de E. Coli)
- Soluciones varias
- Soluciones M9
- Soluciones S basal y completa
- Congelado
- Sincronización con cloro
- Comida para gusanos
- Microscopia
- Datos útiles cant. Gus. p/experimentos
- Cuantificación Proteínas (Bradford)
- Western Blot
- Extraccion de RNA/RTPCR
>> Agar de crecimiento de gusanos (NGM):
| Normal | Rico | Normal | Rico | Normal | |
| NGM (ml) | 60 | 60 | 180 | 180 | 500 |
| ClNa (g) | 0,18 | 0,072 | 0,54 | 0,216 | 1,5 |
| Agar (g) | 1,02 | 1,5 | 3,06 | 4,5 | 8,5 |
| Peptona (g) | 0,15 | 1,2 | 0,45 | 3,6 | 1,25 |
| H2Od (ml) | 58,5 | 58,5 | 175,5 | 175,5 | 487,5 |
Agregar buzo magnético al erlenmeyer y Autoclavar.
Luego de autoclavar, agregar en orden: el buffer PO4 (mezclar), MgSO4 (mezclar), CaCl2 (mezclar) y colesterol (mezclar). NOTA: ojo, hay que hacerlo con el agar aún líquido pero no muy caliente. Si no se mezcla bien antes, el Cacl2 forma cristales.
Agregar: | |||
| para Volumen 60ml | Para volumen 180ml | Para volumen 500ml | |
| Buffer PO4 (ml) | 1,5 | 4,5 | 12,5 |
| MgSO4 1M (ml) | 0,06 | 0,18 | 0,5 |
| CaCl2 (ml) | 0,06 | 0,18 | 0,5 |
| Colesterol | 0,06 | 0,18 | 0,5 |
Volumen NGM necesario para las placas:
Placas chicas: 6 ml
Placas grandes: 18 ml
>> LB-agar de crecimiento de E. coli:
| LB-agar (ml) | 15 | 25 | 30 | 50 | 60 | 75 | 90 | 100 |
| Triptona (g) | 0,15 | 0,25 | 0,30 | 0,50 | 0,60 | 0,75 | 0,90 | 1,00 |
| Extracto de levadura (g) | 0,08 | 0,13 | 0,15 | 0,25 | 0,30 | 0,38 | 0,45 | 0,50 |
| NaCl (g) | 0,08 | 0,13 | 0,15 | 0,25 | 0,30 | 0,38 | 0,45 | 0,50 |
| Agar (g) | 0,23 | 0,38 | 0,45 | 0,75 | 0,90 | 1,13 | 1,35 | 1,50 |
Llevar a volumen con H2Od, ajustar el pH a 7 y autoclavar.
>> Cultivo de E. coli (medio líquido):
| LB (ml) | 15 | 25 | 30 | 50 | 60 | 75 | 90 | 100 | 500 |
| Triptona (g) | 0,15 | 0,25 | 0,30 | 0,50 | 0,60 | 0,75 | 0,90 | 1,00 | 5,00 |
| Extracto de levadura (g) | 0,08 | 0,13 | 0,15 | 0,25 | 0,30 | 0,38 | 0,45 | 0,50 | 2,50 |
| NaCl (g) | 0,08 | 0,13 | 0,15 | 0,25 | 0,30 | 0,38 | 0,45 | 0,50 | 2,50 |
Llevar a volumen con H2Od, ajustar el pH a 7 y autoclavar.
>> Soluciones para los medios de cultivo:
| Buffer fosfato | ||
| Volumen (ml) | 100 | 150 |
| KH2PO4 (g) | 10,83 | 16 |
| K2HPO4 (g) | 3,56 | 5,2 |
| H2O (ml) | 100 | 150 |
Ajustar el pH a 6 con KOH concentrado y autoclavar.
| Sulfato de Magnesio 1M | |
| Volumen (ml) | 10 |
| MgSO4 (g) | 1,2 |
| H2O (ml) | 10 |
Autoclavar
>>Cloruro de Calcio 1M.
| Cloruro de Calcio 1M | |
| Volumen (ml) | 10 |
| CaCl2 (g) | 0,555 |
| H2O (ml) | 10 |
Autoclavar
>>Cloruro de Calcio 1M (a partir del Dihidratado)
| Volumen (ml) | 10 |
| CaCl2.2H2O (ml) | 1,4702 |
| H2O (ml) | 10 |
Autoclavar
>>Colesterol.
| Colesterol (5mg/ml) | |
| Volumen (ml) | 10 |
| Colesterol sigma (g) | 0,05 |
| EtOH 100 % | 10 |
No se autoclava!
>> NaOH
| NaOH 1N | |
| Volumen (ml) | 10 |
| NaOH (g) | 0,4 |
| H2O (ml) | 10 |
No se autoclava!
>> Buffer M9:
| Volumen (ml) | 1000 (1X) | 500 (1X) | 250 (1X) | 1000 (1,25X) |
| KH2PO4 (g) | 3 | 1,5 | 0,75 | 3,75 |
| Na2HPO4 (g) | 6 | 3 | 1,5 | 7,5 |
| NaCl (g) | 5 | 2,5 | 1,25 | 6,25 |
| H2O | 1000 | 500 | 250 | 1000 |
| Después de autoclavar agregar | ||||
| MgSO4 1M | 1 ml | 500 ul | 250 ul | 1,25 |
Buffer M9 partiendo de MgSO4 Solido:
| KH2PO4 (g) | 3 |
| Na2HPO4 (g) | 6 |
| NaCl (g) | 5 |
| MgSO4 1M (g) | 0,12 |
| H2Od | 1000 |
Autoclavar.
>> Preparación de medio S
Medio S basal
– 5,9 gr de ClNa
– 50 ml de fosfato de potasio 1M, pH = 6,0
– 1 ml de colesterol (5mg/ml en EtOH)
– Llevar a 1l con agua destilada
– Autoclavar
Medio S completo
Por cada litro de medio S basal adicionar:
– 10 ml de citrato de potasio 1M pH = 6,0
– 10 ml de trace metals solutions
– 3 ml de CaCl2 1M
– 3 ml de MgSO4 1M
Cada una de estas soluciones se autoclavan por separado.
>> Sincronización con cloro:
Solución de NaOH/Cl2 (Partiendo de cloro 5%)
| Volumen (ml) | 10 | 15 | 20 | 30 | 40 |
| Cloro 5% (ml) | 3,8 | 5,7 | 7,6 | 11,4 | 15,2 |
| NaOH 1N (ml) | 5 | 7,5 | 10 | 15 | 20 |
| H2O (ml) | 1,2 | 1,8 | 2,4 | 3,6 | 4,8 |
Solución de NaOH/Cl2 (Partiendo de cloro 6%)
| Volumen (ml) | 10 | 15 | 20 | 30 | 40 |
| Cloro 6% (ml) | 3,16 | 4,74 | 6,33 | 9,50 | 12,6 |
| NaOH 1N (ml) | 5 | 7,5 | 10 | 15 | 20 |
| H2O (ml) | 1,84 | 2,76 | 3,67 | 5,5 | 7,4 |
Protocolo de sincronización:
- Lavar las placas (con gusanos con muchos huevos en la panza) con M9 aspirando y botando con pipeta (P1000). El producto lavado se pone en falcon de 15 ml.
- Centrifugar para pelletear los gusanos (en centrífuga ROLCO: 20seg a velocidad media). Eliminar el sobrenadante.
- Agregar 5 ml de la solución de NaOH/Cl2 y agitar suavemente con la mano por 4 minutos (exactos).
- Agregar 2 volúmenes de M9 estéril (hasta arriba del tubo) para diluir la solución de cloro/NaOH. Con este tratamiento se rompen los gusanos y se desprenden los huevos. Se eliminan bacterias.
- Centrifugar y eliminar el sobrenadante (en centrífuga ROLCO: 20seg a velocidad media).
- Realizar dos lavados con M9 estéril (hasta arriba del tubo). Centrifugar entre ambos lavados.
- Los huevos se resuspenden en buffer M9 (Volumen final 3,5ml + 35ul de ATB 100X) y se crecen en agitación (110rpm) ON. Bajo esas condiciones los huevos eclosionan a larva L1 y permanecen en ese estadio larval hasta que se les dé de comer.
Los huevos sincronizados también se pueden colocar en placas con o sin comida, dependiendo del experimento. Los huevos se ponen en placas sin comida para que eclosionen las L1 y no puedan seguir creciendo (quedan sincronizadas en éste estadio luego de un día). Al día siguiente las L1 se ponen en una placa con comida y quedan sincronizadas.
>> Congelado
Congelado de gusanos en freezing solution:
- Lavar con 1ml de Freezing solution cada placa de agar mediano con muchos L1.
- Alicuotar 500ul en cada criotubo. Congelar a -80°C en caja de tergopol.
Soluciones:
Freezing solution: 50% M9 + 50% Buffer B.
| Buffer B | 1000ml | 50ml | 30ml |
| NaCl (g) | 5.7 | 0,285 | 0,171 |
| KH2PO4 1M pH = 6 (ml) | 50 | 2.5 | 1.5 |
| Glicerol(ml) | 300 | 15 | 9 |
| H2Od (ml) | 644 | 32.2 | 19.32 |
Ajustar a pH = 6 y autoclavar. Luego de autoclavar agregar 0,3ml de MgSO4 1M (esteril).
Preparación de 5ml de MgSO4 1M:
1mol MgSO4 ………. 120,37gr
1M = 120,37gr/1000ml*5ml = 0,6 gr/5ml
Preparación de KH2PO4:
1M …….. 136,1gr/l = 6,8gr/50ml = 0,34gr/2,5ml
Descongelado: Sacar de -80°C las muestras y dejar en la mesada hasta que los bordes se derritan. Tirar todo el contenido del tubo en una placa con agar y E. Coli.
Congelado de gusanos en Soft agar:
- Derretir el Soft agar en baño a 50°C o microondas.
- Usar una placa grande (10cm) o 2-3 placas medianas con gusanos en estadío L1.
- Lavar con 0,6ml de S Buffer las placas. Pasar a un tubo estéril y mantener 15 minutos en hielo.
- Agregar igual volumen de agar y mezclar bien.
- Alicuotar 1ml a un criotubo y congelar a -80°C en caja de tergopol.
Soluciones:
S buffer: K2HPO4 0,05M …………. 129ml
KH2PO4 0,05M …………. 871ml
NaCL ………………………… 5,85gr
pH = 6, autoclavar.
Soft agar: NaCL ………………………. 0,58gr
KH2PO4 ………………….. 0,68gr
K2HPO4 ………………….. 0,87gr
Glycerol …………………… 30ml
Agar ………………………… 0,4gr
H2O ………………………… 100ml
pH = 6, autoclavar.
Descongelado:
- Sacar el vial de -80°C y colocarlos en una caja con hielo (trabajar con rapidez para evitar que se descongele).
- Flamear una espátula. Remover 0,25ml del vial.
- Congelar el resto del vial nuevamente a -80°C.
- Pasar los gusanos de la espátula a una placa con agar y E. coli.
Congelado de bacterias:
- A 500ul de cultivo LB con las bacterias agregar 88ul de glicerol 100% estéril.
- Congelar a -80°C.
Descongelado: Sacar de -80°C los tubos. Dejar descongelar e inocular con ésta suspensión.
>> Comida para gusanos:
OP50 muertas: esto sirve para extracción de RNA, proteínas y experimentos de estrés.
· Preparar 3 erlenmeyer con 500ml de medio LB c/u.
· Sembrar cada erlenmeyer con 200ul de bacteria (OP50).
· Crecer ON a 37°C y 200 rpm.
· Centrifugar en falcon de 50ml durante 10 minutos a 5000 rpm.
· Descartar el sobrenadante (guardar algunos falcon con pellet sin tratamiento con N2 líquido).
· Matar el resto de las bacterias con N2 líquido.
· Pasar los falcon por H2O a 50°C (de la canilla).
· Agregar el doble de volumen de M9.
· Vortexear.
· Tomar una alícuota, hacer una dilución 1/250 o 1/500 de la misma y medir DO600.
· Almacenar a -20°C.
Usar para cultivo: máx. DO600 = 1,0 final (15 gusanos/20ul).
Comida para gusanos (placas grandes):
· Preparar 1 erlenmeyer con 500ml de medio LB.
· Sembrar 200ul de bacteria (OP50).
· Crecer ON a 37°C y 200 rpm.
· A cada placa grande se agrega 200ul del cultivo ON.
· Rastrillar.
· Dejar secar las placas.
· Almacenar a 4°C.
Comida para gusanos (placas chicas):
· Preparar 1 erlenmeyer con 500ml de medio LB.
· Sembrar 200ul de bacteria (OP50).
· Crecer ON a 37°C y 200 rpm.
· A cada placa chica agregar 5 gotas del cultivo ON distribuidas por la placa.
· Dejar secar las placas.
· Almacenar a 4°C.
>> Microscopía:
Fijación de gusanos GFP (metanol/acetona):
· Tomar muestras de gusanos (20ul del erlenmeyer) en eppendorf de 1,5ml.
· Remover el exceso de líquido por centrifugación (1 min. en centrífuga de células, 1500 rpm aproximadamente).
· Congelar rápidamente poniendo el eppendorf en isopropanol a -80°C mantenido en recipiente con hielo y sal (cracking por frío para permeabilizar la cutícula y que puedan fijarse mejor).
· Agregar 100ul de metanol -20°C.
· Incubar por 5 min. en agitación.
· Centrifugar. Remover el metanol.
· Agregar 10ul de acetona -20°C.
· Incubar 5 min. en agitación.
· Centrifugar. Remover la acetona.
· Rehidratar los gusanos en etanol 95% por 2 min.
· Remover el alcohol por centrifugación.
· Rehidratar los gusanos en etanol 75% por 2 min.
· Remover el alcohol por centrifugación.
· Rehidratar los gusanos en etanol 50% por 2 min.
· Remover el alcohol por centrifugación.
· Rehidratar los gusanos en etanol 30% por 2 min.
· Remover el alcohol.
· Montarlos.
Montaje:
Medio especial: Microscopía confocal
· 5ul DAKO + 1,5ul de gusanos.
Económico: Dura 3 días
· 4ul glicerol 40% + 1,5ul de gusanos.
Poner cubre y sellar con esmalte de uñas.
Tiempo previsto 23 minutos.
Fijación de gusanos inmuno fluorescentes (paraformaldehído):
Solución stock de fijación:
· 20% paraformaldehído: 250mg.
· Formaldehído en 1,125ml de NaOH 5mM.
Incubar 30 min. aproximadamente a 65°C para disolver (preparar solución fresca antes de usar).
Protocolo:
· Lavar las cepas de C. elegans en M9, PBS o H2O en tubos de 15ml varias veces. Durante los lavados preparar solución de fijación.
· Agregar aproximadamente 1ml de solución de fijación (4% paraformaldehído) a los gusanos en tubos de 15ml o eppendorf.
· Incubar 24hs a 4°C.
Protocolo modificado para estudios circadianos (mucho background):
· Tomar 20ul de gusanos en estadío L1 de erlenmeyer sincronizados por 4 días ( más de 50 gusanos). Usar eppenorf de 1,5ml. Centrifugar y quitar buffers.
· Agregar 100ul de paraformaldehído 20%. Incubar 15min. a 4°C.
· Diluir con 400ul de PBS pH 0 7,4. Incubar ON a 4°C.
· Centrifugar 15”. Remover el buffer y lavar en 1ml de PBS pH = 7,4.
· Centrifugar 15”. Remover el buffer. Tomar 1,5ul de gusanos y montar en medio DAKO.
Anestesia (para mirar in vivo en microdcopía):
- Azida 10% solución stock.
- Dilución: se agrega 3,5ul de azida stock en 700ul de H2O.
Se utilizarán 3,5ul de esta solución para 20ul de gusanos en M9.
>> Cuenta de gusanos para experimentos:
Máximos volúmenes:
- 35ml en erlenmeyers de 250ml.
- 7ml en erlenmeyers de 50ml.
Por sincronización de placas grandes (100mm) de TJ1060 se obtienen 22.000 gusanos aproximadamente. Se crecen a 18°C. Para el experimento se lleva a 25,3°C.
Experimento de alimentación:
- Se usan cultivos de 6gusanos/20ul. Se toman muestras de 50ul por punto.
- Muestras por duplicado.
Experimento de ritmo shock:
- Se usan alícuotas de 20 gusanos/20ul
- Se parte de E. coli con Abs (600) = 1,0 (dil = 1/50).
Se toman 20ul de gusanos + 30ul de solución (NaCL o H2O2) y se deja por dos horas en well de 96. Por triplicado cada punto más un control. Muestras cada 6 horas.
Experimento de extracción de RNA:
- Se usan cultivos de 30 gusanos/20ul máximo.
Se toman 2ml de cultivo (3000 gusanos) → con 400ul de Trizol. Se obtienen aproximadamente = 25ug de RNA total (9ng/gusano aproximadamente).
Extracción de proteína:
- Se usan cultivos de 30gusanos/20ul máximo.
Proteínas 200ul * 2ug/ul = 400ug de proteínas en 2.150 gusanos (homogeneizados en 100ul de buffer fosfato y llevado a volumen finas = 200ul).
>> Cuantificación de Proteínas – Bradford:
Bio-Rad Protein Assay
- Preparar el reactivo diluyendo una parte del concentrado en 4 aparte de agua desionizada. Se conserva a temperatura ambiente hasta 2 semanas.
- Preparar diluciones de la proteína estándar (BSA), la cual es representativa de la solución proteica a testear. El rango lineal de éste método es 0,05mg/ml a 0,5mg/ml aproximadamente.
- Pipetear 10ul de cada solución estándar o la muestra en cada well de una placa de 96 well. Realizar duplicados para cada muestra.
- Agregar 200ul del reactivo diluido a cada well. Mezclar bien la muestra con el reactivo utilizando la micro pipeta. Cambiar el tip para agregar el reactivo y mezclar en cada well.
- Incubar a temperatura ambiente por 5 minutos por lo menos, hasta no más de una hora.
- Medir absorbancia a 595nm en un lector de micro placas.
| BSA 1ug/ul (ul) | 10 | 15 | 20 | 25 | 30 | 35 | 40 | 45 | 50 |
| H2O (ul) | 90 | 85 | 80 | 75 | 70 | 65 | 60 | 55 | 50 |
| Volumen final (ul) | 100 | 100 | 100 | 100 | 100 | 100 | 100 | 100 | 100 |
| BSA [ug/ul] | 0,1 | 0,15 | 0,2 | 0,25 | 0,3 | 0,35 | 0,4 | 0,45 | 0,5 |
Diluir las muestras 1/10.
Western blot
1- Realizar un gel de poliacrilamida (el porcentaje del gel depende de la proteína a separar).
2- Incubar el gel con el buffer de transferencia, 15minutos a temperatura ambiente en agitación.
3- Transferencia del gel a la membrana: transferir 30minutos a 10volts.
4- Teñir la membrana con rojo Ponceau: 10 minutos en agitación.
5- Lavar la membrana con agua destilada: a través de la coloración con rojo ponzeau visualizamos las bandas en la membrana. Escanear. Marcar las bandas del marcador de masa. Si se van a usar anticuerpos secundarios diferentes en este paso se debe cortar la membrana.
6- Bloquear la membrana con leche descremada en polvo, 1h a temperatura ambiente. Se usan aproximadamente 30ml de leche 10%P/V por membrana. Para diluir la leche en polvo se usa buffer TTBS.
7- Realizar 3 lavados de 10min cada uno con TTBS, en agitación y a temperatura ambiente.
8- Incubar la membrana con el anticuerpo primario. La membrana entera lleva 3ml de anticuerpo. Este paso hay que ponerlo a punto: 1) incubar durante 1h a temperatura ambiente y en agitación o 2) incubar ON en la cámara fría en agitación. Para melanopsina usar una dil 1/1000 del anticuerpo. Para la dilución usar buffer TTBS.
9- Realizar 3 lavados de 10min cada uno con TTBS, en agitación y a temperatura ambiente.
10- Incubar la membrana con el anticuerpo secundario, 2hs a temperatura ambiente y en agitación. Preparar una dil 1/5000 en TTBS.
11- Realizar 3 lavados de 10min cada uno con TTBS, en agitación y a temperatura ambiente.
12- Revelar:
Solución 1:
-100ul luminol (2,5mM)
– 44ul de p-coumaric acid (400uM)
– 1ml de tris-HCl 1M pH=8,5 (100mM)
-H2O a 10ml
Alicuotar en fracciones de 1ml.
Solución 2: para preparar 1ml (se prepara en el momento)
-0,61ul de H2O2 (5,4mM)
– 100ul de tris-HCl 1M pH=8,5 (100mM)
– 900ul de H2O
Mezclar 1ml de la solución 1 con 1ml de la solución 2 (en el cuarto oscuro). Agregar la mezcla de a 500ul sobre la membrana. Dejar actuar 5min. Colocar la membrana en el casette de revelado y encima colocar la placa de revelado cortada previamente (el tiempo de exposición se puede variar: 1, 2 o 3min, para melanopsina). Colocar la placa en la solución de revelado y luego en la solución de fijación. Finalmente colocar las placas reveladas en agua de la canilla.
Stripping
1- Incubar la membrana con el buffer de stripping (agregarle β-mercaptoetanol: por cada 100ml de buffer agregar 700ul de β-mercaptoetanol), 30minutos a 50ºC en agitación.
2- Teñir la membrana con rojo Ponzeau: 10 minutos en agitación.
3- Lavar la membrana con agua destilada.
4- Bloquear la membrana con leche descremada en polvo, 1h a temperatura ambiente. Se usan aproximadamente 30ml de leche 10%P/V por membrana. Para diluir la leche en polvo se usa buffer TTBS.
5- Realizar 3 lavados de 10min cada uno con TTBS, en agitación y a temperatura ambiente.
6- Incubar la membrana con el anticuerpo secundario, 2hs a temperatura ambiente y en agitación. Preparar una dil 1/5000 en TTBS.
7- Realizar 3 lavados de 10min cada uno con TTBS, en agitación y a temperatura ambiente.
8- Revelar.
>> Protocolo de síntesis 1er cadena cDNA:
Experimentos de ritmos TJ1060:
Toma de Muestras:
A tener en cuenta:
2000gusan = 16ug de RNA total (9ng/worm aprox)
Rotular eppendorf a usar de 2ml con marca con espátula caliente (el fibron se borra)
PROTOCOLO:
– Tomar 2ml gusanos [1 a 1.25gus/ul] y pasar a eppendorf de 2ml.
– Pelletear a 1600rpm 1.5minutos centrifuga eppendorf.
– Descartar sobrenadante con pipeta P1000.
3x lavados:
– Agregar 2ml de M9esteril, resuspender con el dedo.
– Pelletear a 1600rpm 1.5minutos centrifuga eppendorf.
– Descartar sobrenadante con pipeta P1000.
– Agregar 200ul trizol.
– Vortex 1’ al máximo.
– Congelar sumergiendo eppendorf en falcon de 50ml con Alcohol isopropilico que se encuentre a -80~C
Al tomar la siguiente muestra sacar el anterior del alcohol isopropilico y meter en bolsa sellada a -80~C. Máximo de eppendorfs que entran por falcon de 50ml con isopropanol = 2
Una vez tomadas TODAS las muestras: Extracción de RNA:
DIA 1
MATERIAL NECESARIO:
– Pote de helado de ¼ con 2 cucharadas de Nitrógeno liquido, tapado.
– Jeringas de tuberculina: 1 por muestra.
– Rotulador y papel para secar el isopropilico antes de rotular.
– Vortex
– Centrifuga de eppendorf a 4~C (meter destapada en heladera 30min antes)
– Trizol: 100ul adicionales por muestra
– CLOROFORMO:ISOAMILICO 49:1 cant 60ul por muestra.
– Isopropanol para uso molecular a -20~C: 150ul aprox por muestra
– EtOH 75% para uso molecular preenfriado a -20~C.
– dH2O-DEPC cant. 12ul por muestra
– Eppendorf de 500ul estériles rotulados igual que muestras (por duplicado)
– Rack (tipo tapa de tacho de helado de telgopor agujereada) para meter los eppendorf en el Nitrogeno (tiene que entrar en el recipiente)
– Adaptadores para centrifugar los eppendorf de 500ul en la centrifuga.
– Tips varios estériles.
– Guantes y antiparras
– mechero
A tener en cuenta: posicionar siempre los eppendorf en la centrifuga todos de la misma forma a fin de saber de que lado queda el pellet por mas que este no se vea.
TIEMPO ESTIMADO: 3horas
PROTOCOLO: Todo cerca de mechero (ojo q los solventes son inflamables)
– Sacar la bolsa con las muestras y rotularlas (en hielo). Lo mas rápido posible.
– Agregar 100ul más de Trizol a cada muestra. (Vf=300ul).vortexear.
– Pasar a eppendorf de 500ul.
– Poner los eppendorf en rack de telgopor
– Congelar en Nitrogeno líquido y descongelar a 37ºC (en ciclador) x 3veces (agitando en vortex a cada rato que va descongelando)
– Homogeneizar con jeringa de tuberculina 10 veces (o sea 10 pasadas). (cambiar la jeringa en cada muestra). OJO: USAR GUANTES, ANTIPARRAS Y MANGA LARGA. EL TRIZOL QUEMA.
– Dejar a Tº ambiente 7,5 minutos.
– Agregar 54 ul de solucion cloroformo:isamilico 49:1.
– Vortexear
– Incubar 2min a Tº ambiente.
– Centrifugar 15’ a 14000rpm 4ºC.
– Mantener tubos en hielo para que las fases no se junten.
– Transferir fase acuosa (la de arriba, Vol maximo 350ul) a otro tubo(eppendorf de 500ul). Usar P200.
– Agregar 150ul isopropanol a la fase acuosa y agitar c/vortex.
– Dejar a –20C al menos 1hora (ON a -80C funciona bien).
– Centrifugar 10’ a 14000rpm 4ºC.
– Remover sobrenadante con pipeta.
– Agregar 300ul de EtOH 75% despegar el pellet sin resuspender.
– Centrifugar 5’ a 14000rpm x 4ºC.
– Remover EtOH con pipeta P200 del otro lado del pellet.
– Secar dado vuelta en papel. air dry 5 a 10min.
– Resuspender en H2O-DEPC. 12ul con pipeta P10.
DIA2
CUANTIFICACION Y CALIDAD DEL RNA:
CUANTIFICACION:
– Determinar la concentración de RNA x espectrofotometría tomando 1ul de RNA y llevándolo a 200ul con dH2O estéril (dilución 1:200). Medir la absorbancia a 260 y 280 (cubetas para RNA de poco volumen); calcular la razón A260/A280 (ideal >1.6 si es menor hay contaminación con proteínas).
– Calcular [] como:
(A260* 40ug/ml * 200(fact dilución) ) * 1ml/1000ul = Xug RNA/ul
– guardar el RNA con todos los datos a –80ºC.
– Anotar todos los datos super bien.
CALIDAD: GEL p/RNA
Calculo para cuba grande 100ml(en caso de usar cuba chica calcular volumen=40ml)
– 1g de Agarosa en 82ml de Buffer 1xMOPS. Calentar microondas.
-esperar que se enfrie un poco y agregar 18ml de Formaldehído.
* Usar buffer 1x MOPS en cuba.
* Sembrar 1.5ug de RNA por calle (agregar loading buffer 3x para RNA)
* Poner PM DNA BsteII 7ul
Correr gel hasta abajo en cuba chica.
Luego de correr el gel teñir con 50mlTBE+5ml SYB (Sybergreen tipo II) incubando por 1hora con agitación en tachito de jabonera (tapado de la luz).
Mirar en transiluminador y sacar foto. Deben verse las bandas ribosomales de C. elegans (2,8Kpb y 1,8kpb), posible contaminación con Coli (2,3kpb y 1,6kpb) y poco chorreado (puede que se vea mancha debajo de todo correspondiente a tRNA).
Ejemplo:
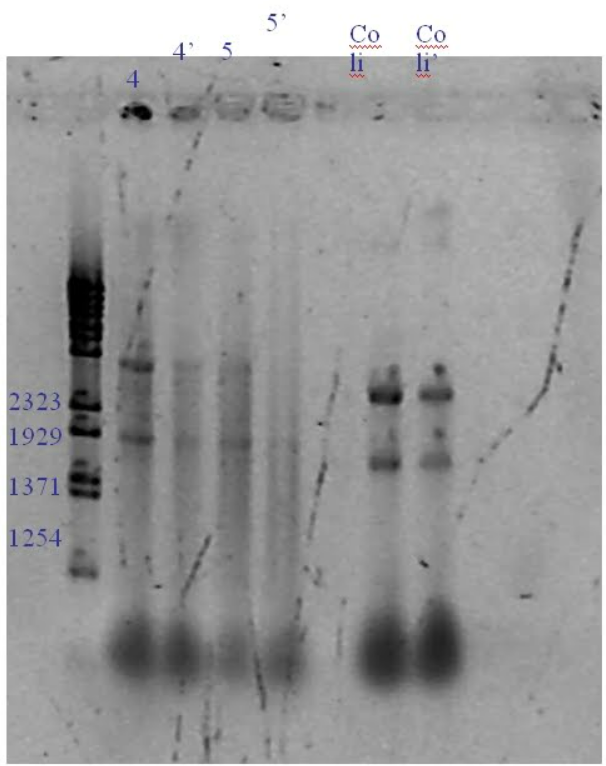
En cuba chica. Peine de 13 calles.
Muestras 4 y 5 extracción RNA gusanos.
Muestras Coli ejemplo de Ribosomal bacteriano.
DIA 3
Sintesis de 1er cadena cDNA:
Se parte de 5ug de RNA (en dH2O-DEPC). (en caso extremo, minimo = 2,5ug)
1) Tratamiento con DNAsa
La DNAsa debe ser inactivable por calor. Este paso puede obviarse si los primers están diseñados para levantar el cDNA y no DNA genómico.
Xul dH2O
Xul RNA (5ug)
1ul DNAsa Buffer 10x
1ul DNAsaI
——————-
10ul final
– incubar 15’ a 25ºC
– Inactivar 10’ a 65ºC.
– Spin down (para bajar la condensación de las paredes)
– Pasar a hielo.
2) Hibridación de oligo-dT
– a los 10ul de la reacción anterior se agrega:
1ul oligo-dT (500ug/ml), incubación 5’a 65ºC (ciclador)
preparar la MIX de RT mientras se espera la incubación.
– pasar a hielo
– esperar al menos 1’ antes de poner la mix de RT (si es mas, mejor)
3)Mix de RT:
| Para 1x | ||||
| 5x reaction Buffer | 2ul | |||
| dNTPs 10mM | 1ul | |||
| dH2O-DEPC | 3ul | |||
| DTT 0.1M | 2ul |
——————–
8ul MIX
Agregar a los 11ul de la reacción anterior (oligodt cDNA)
– poner en ciclador 5’ a 37ºC
– Añadir 1ul de MMLV-RT (Transc. Reversa) = 20ul final de reacción
– 37ºC 1hora ß OJO!!!!! Depende de la enzima. Algunas son a 42ºC!!!
– inactivar 70ºC 15’
– tratamiento con RNAsaH: 10’ a 37ºC (si no se trata anda igual para amplific hasta 1k)
– spin down
* Luego de cada mezcla dar vortex y spin corto en centrifuga de células.
– Guardar a –20ºC
